Çok türlü birleşme süreci - Multispecies coalescent process
Çok Türlü Birleşme Süreci , birkaç türden alınan bir DNA dizisi örneği için soy ilişkilerini tanımlayan stokastik bir süreç modelidir . Birden fazla tür durumunda birleştirici teorinin uygulanmasını temsil eder . Çok türün birleşmesi, tek bir gen için türler arasındaki ilişkilerin ( gen ağacı ) türün daha geniş geçmişinden ( tür ağacı ) farklı olabileceği durumlarda sonuçlanır . Filogenetik teori ve pratiği ve genom evrimini anlamak için önemli çıkarımları vardır .
Bir gen ağacı , yeniden birleşmeyen bir lokus için bir dizi örneği arasındaki evrimsel ilişkileri tanımlayan ikili bir grafiktir. A s PECIES ağaç evrim ağaç gibi varsayarak, türlerin bir dizi arasındaki evrimsel ilişkileri açıklar. Bununla birlikte, birkaç süreç, gen ağaçları ve tür ağaçları arasında uyumsuzluğa yol açabilir . Multispecies Coalescent modeli atalarının için gerekli hesaplamaların türler filogenezlerini çıkarım için bir çerçeve sağlar polimorfizmi ve gen ağaç türlerinin ağaç çatışma. İşlem ayrıca Sansürlü Birleşme olarak da adlandırılır .
Tür ağacı tahmininin yanı sıra, çoklu türlerin kaynaşma modeli, türlerin farklılaşma sürelerinin tahmini, ata türlerin popülasyon büyüklükleri, türlerin sınırlandırılması ve türler arası gen akışının çıkarımı gibi bir dizi biyolojik sorunu ele almak için genomik verileri kullanmak için bir çerçeve sağlar. .
Gen ağacı-tür ağaç uyumu

En basit, önemsiz olmayan filogenetik ağaç olan köklü bir üç takson ağacını ele alırsak, üç farklı ağaç topolojisi, ancak dört olası gen ağacı vardır. Daha az sayıda topolojiye rağmen dört farklı gen ağacının varlığı, birleşme zamanlarında farklılık gösteren topolojik olarak özdeş gen ağaçlarının olduğu gerçeğini yansıtmaktadır. Tip 1 ağaçta, A ve B türlerindeki aleller, AB soyunu C soyundan ayıran türleşme olayından sonra birleşir. Tip 2 ağaçta, A ve B türlerindeki aleller, AB soyunu C soyundan ayıran türleşme olayından önce birleşir (başka bir deyişle, tip 2 ağaç derin bir birleşme ağacıdır). Tip 1 ve tip 2 gen ağaçlarının her ikisi de tür ağacıyla uyumludur. Diğer iki gen ağacı tür ağacından farklıdır; iki uyumsuz gen ağacı da derin birleşme ağaçlarıdır.
Bütün bu ağaçlar için birleşme zamanlarının dağılımı aslında süreklidir. Başka bir deyişle, aynı gen ağacına sahip herhangi iki lokus için tam birleşme süresi farklı olabilir. Bununla birlikte, birleşmenin en erken türleşme olayından önce mi sonra mı gerçekleştiğine bağlı olarak ağaçların parçalanması uygundur.
Birleştirilmiş birimlerdeki iç dal uzunluğu göz önüne alındığında, her bir gen ağacının olasılığını hesaplamak kolaydır. Diploid organizmalar için birleşik birimlerdeki dal uzunluğu, türleşme olayları arasındaki nesil sayısının etkin popülasyon boyutunun iki katına bölümüdür. Derin birleşme ağacının üçü de eşit olasılıklı olduğundan ve bu derin birleşme ağacından ikisi uyumsuz olduğundan, köklü bir üç taksonlu gen ağacının tür ağacıyla uyumlu olma olasılığını görmek kolaydır:
Birleşik birimlerdeki dal uzunluğunun ( T ) alternatif bir biçimde yazıldığı yerde: nesil sayısı ( t ) bölü etkin popülasyon büyüklüğünün ( N e ) iki katı . Pamilo ve Nei ayrıca, dört ve beş taksonun köklü ağaçları için uyum olasılığını ve daha büyük ağaçlar için uyum olasılığının genel bir üst sınırını da türetmiştir. Rosenberg, topolojilerin tamamı için kullanılan denklemleri takip etti (takson sayısı arttıkça mümkün hale gelen çok sayıda farklı filogenetik ağaç, takson sayısı çok sınırlı olmadığı sürece bu denklemleri pratik yapmaz).
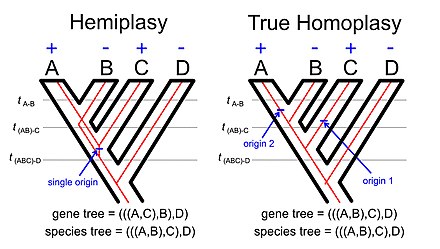
Hemiplazi fenomeni, gen ağacı-tür ağaç uyumsuzluğunun altında yatan temel fikrin doğal bir uzantısıdır. Tür ağacıyla uyuşmayan bazı karakterlerin dağılımını düşünürsek, bu homoplastiyi (karakterin çoklu bağımsız kökenleri veya tek bir köken ve ardından çoklu kayıplar) yansıtabilir veya hemiplaziyi (özelliğin tek bir kökeni ile ilişkili olan) yansıtabilir. tür ağacıyla uyuşmayan bir gen ağacı).
Fenomeni olarak adlandırılır eksik soy sıralama (genellikle bilimsel literatürde ILS kısaltılmıştır) olgusuna bağlıdır. Köklü dört taksonlu bir ağaç kullanarak hemiplazi örneğini incelersek (sağdaki resme bakın), A, B ve C taksonlarının ortak ataları ile A ve B taksonlarının ortak ataları arasındaki soy polimorfik olmalıdır. türetilmiş özelliğe sahip alel (örneğin, yeri değiştirilebilen bir eleman yerleştirme) ve atasal özelliğe sahip alel. Eksik soy sıralama kavramı, nihayetinde bir veya daha fazla türleşme olayı boyunca polimorfizmlerin kalıcılığını yansıtır.
Çoklu türlerin birleştirilmesinin matematiksel açıklaması
Gen ağaçlarının çoklu tür birleştirici modeli altındaki olasılık yoğunluğu, çoklu lokus dizi verileri kullanılarak parametre tahmini için kullanımı ile birlikte tartışılmaktadır.
varsayımlar
Temel çoklu türlerin birleştiği modelde, türlerin soyoluşunun bilindiği varsayılır. Türlerin ayrılmasından sonra, göç, hibridizasyon veya introgresyon olmadan tam izolasyon da varsayılır. Lokus içindeki tüm sitelerin aynı gen ağacını (topoloji ve birleşme süreleri) paylaşacak şekilde rekombinasyon olmadığını varsayıyoruz. Bununla birlikte, temel model, göç veya introgresyon, popülasyon büyüklüğü değişiklikleri, rekombinasyona uyum sağlamak için farklı şekillerde genişletilebilir.
Veri ve model parametreleri
Bu yöntemin modeli ve uygulaması herhangi bir tür ağacına uygulanabilir. Örnek olarak büyük maymunların tür ağacı : insanlar (H), şempanzeler (C), goriller (G) ve orangutanlar (O) ele alınmaktadır. Tür ağacının (((HC)G)O)) topolojisinin, analizde bilindiği ve sabitlendiği varsayılır (Şekil 1). Izin tüm veri setini olmak lokusunda dizi sıralaması temsil sahip, toplam loci.
Mevcut bir türün popülasyon büyüklüğü, yalnızca o türden bazı lokuslarda birden fazla birey örnekleniyorsa dikkate alınır.
Şekil 1 'deki örneğin bir model parametreleri üç ıraksama kez dahil , ve ve popülasyon boyutu parametreleri insanlar için; şempanzeler için; ve , ve üç ata türü için.
Iraksama süreleri ( 'ler), tür ağacındaki ata düğümünden günümüze kadar site başına beklenen mutasyon sayısı ile ölçülür (Şekil 1, Rannala ve Yang, 2003).
Bu nedenle, parametreler .
Gen soylarının dağılımı
Ortak dağılımı doğrudan bu bölümde elde edilir. Farklı türlerden iki dizi, yalnızca iki türün atası olan bir popülasyonda birleşebilir. Örneğin, H ve G dizileri, HCG veya HCGO popülasyonlarında birleşebilir, ancak H veya HC popülasyonlarında birleşemez. Farklı popülasyonlardaki birleşme süreçleri farklıdır.
Her populasyon için şecere, zaman içinde geriye doğru, populasyonun sonuna kadar izlenir ve populasyona giren soy sayısı ve onu terk eden soy sayısı kaydedilir. Örneğin, ve H popülasyonu için (Tablo 1). Bu sürece sansürlü birleşme süreci denir, çünkü bir popülasyon için birleşme süreci, popülasyona giren tüm soylar birleşmeden önce sonlandırılabilir. Eğer nüfus oluşur bağlantısız alt ağaçlar veya soyları.
Bölge başına bir mutasyon biriktirmek için geçen süre olarak tanımlanan bir zaman birimi ile, herhangi iki soy, oranda birleşir . Bekleme süresi gelen soylar sayısını azaltır sonraki birbirine yakınlaşmış olaya kadar hiç üstel yoğunluğu
Eğer sonuncusu ile popülasyonun sonu arasında herhangi bir birleşme olayının meydana gelmeme olasılığı ise ; yani zaman aralığı sırasında . Bu olasılık 1 ise ve 1'dir .
(Not: Bir zaman aralığında hiçbir etkinlik olasılığını hatırlayalım oranına sahip bir Poisson süreci için ise varken birbirine yakınlaşmış oranını Buraya. Soyları edilir .)
Ek olarak, popülasyondaki belirli bir gen ağacı topolojisinin olasılığını elde etmek için, bir soy örneğinde bir birleşme olayı meydana gelirse, belirli bir soy çiftinin birleşme olasılığı .
Bu olasılıkların çarpılmasıyla , gen ağacı topolojisinin popülasyondaki ortak olasılık dağılımı ve birleşim süreleri aşağıdaki gibi çarpılır:
- .
Lokus için gen ağacının ve birleşme zamanlarının olasılığı, tüm popülasyonlardaki bu tür olasılıkların ürünüdür. Bu nedenle, Şekil 1'deki gen şeceresine sahibiz.
Olasılığa dayalı çıkarım
Her lokustaki gen şeceresi , ağaç topolojisi ve birleşme zamanları ile temsil edilir . Tür ağacı ve üzerindeki parametreler göz önüne alındığında, birleşme işlemi ile olasılık dağılımı şu şekilde belirlenir:
- ,
locus locus'taki gen ağacının olasılık yoğunluğu nerede ve ürün, gen ağaçlarının parametrelerden bağımsız olduğunu varsaymamızdır.
Lokusta gen ağacı ve birleşme süreleri (ve dolayısıyla dal uzunlukları) verilen verilerin olasılığı , Felsenstein'ın filogenetik olasılığıdır. Lokuslar arasında bağımsız evrim varsayımı nedeniyle,
Parametreler verilen dizi verilerinin olabilirlik fonksiyonu veya olasılığı , gözlemlenmemiş gen ağaçlarının ortalamasıdır.
burada entegrasyon, tüm olası gen ağacı topolojileri ( ) üzerindeki toplamı ve her lokustaki her olası topoloji için, birleşme zamanları üzerindeki entegrasyonu temsil eder . Bu, çok küçük tür ağaçları dışında genel olarak inatçıdır.
In Bayes çıkarsama , biz parametreleri üzerinde önceden atamak ve sonra arka olarak verilmektedir
burada yine entegrasyon, tüm olası gen ağacı topolojileri ( ) üzerindeki toplamı ve birleşme zamanları üzerindeki entegrasyonu temsil eder . Uygulamada, gen ağaçları üzerindeki bu entegrasyon , parametrelerin ve gen ağaçlarının ortak koşullu dağılımından örnekler alan bir Markov zinciri Monte Carlo algoritması ile gerçekleştirilir.
Yukarıdakiler, tür ağacının sabit olduğunu varsayar. Tür ağacı tahmininde, tür ağacı ( ) da değişir, böylece ortak koşullu dağılım (MCMC örneklerinden gelen)
tür ağaçlarda önceki nerede .
İki aşamalı özet yöntemlerinden önemli bir sapma olarak, tam olabilirlik yöntemleri, gen ağaçlarının ortalamasıdır. Bu, gen ağaçlarındaki dal uzunluklarındaki (birleşme süreleri) bilgiyi aynı anda kullandıkları ve belirsizliklerini (hizalamalardaki sınırlı dizi uzunluğundan dolayı) aynı anda barındırdıkları anlamına gelir. Ayrıca, tam olabilirlik yöntemlerinin neden iki aşamalı özet yöntemlerinden hesaplama açısından çok daha fazla talep ettiğini de açıklıyor.
Markov zinciri Monte Carlo, çoklu türlerin birleşmesi altında
Yukarıdaki olabilirlik fonksiyonunun tanımındaki gen ağaçları üzerindeki entegrasyon veya toplamı, sadece iki veya üç türe sahip çok küçük tür ağaçları dışında hesaplamak neredeyse imkansızdır. Dizi hizalamalarında olabilirlik fonksiyonunun hesaplanmasına dayanan tam olabilirlik veya tam veri yöntemleri, bu nedenle çoğunlukla Markov zinciri Monte Carlo algoritmalarına güvenmiştir. Çok türlü birleştirici model altındaki MCMC algoritmaları, Bayesci filogenetikte kullanılanlara benzerdir, ancak esas olarak çoklu lokustaki gen ağaçlarının ve tür ağacının uyumlu olması gerektiğinden belirgin şekilde daha karmaşıktır: dizi farklılığı türlerden daha eski olmalıdır. uyuşmazlık. Sonuç olarak, gen ağaçları sabitken tür ağacının değiştirilmesi (veya tür ağacı sabitken bir gen ağacının değiştirilmesi), zayıf karıştırma özelliklerine sahip verimsiz algoritmalara yol açar. Tür ağacını ve gen ağaçlarını koordineli bir şekilde değiştiren akıllı algoritmalar tasarlamak için önemli çabalar sarf edilmiştir; türlerin farklılaşma sürelerini, koordineli NNI, SPR ve NodeSlider hareketlerini değiştirmek için lastik bant algoritmasında olduğu gibi.
Örneğin, iki türünün (bir durum göz önünde bir ve B bir sekans sapmasına kez) ve her bir mahalde iki dizi, lokusunda . Biz herkes için . Akımın kısıtlaması dahilinde tür sapma zamanını değiştirmek istediğimizde, en küçüğüyle neredeyse aynı olabileceğinden , değişim için çok az yerimiz olabilir . Lastik bant algoritması , dikkate alınmadan değişir ve daha sonra , lastik bant bir uca doğru çekilen sabit bir noktadan tutulduğunda bir lastik bant üzerindeki işaretlerin hareket ettiği şekilde deterministik olarak değiştirir . Genel olarak, lastik bant hareketi, gen ağaçlarındaki düğümlerin yaşlarının, değiştirilmiş türlerin ayrışma süresiyle uyumlu kalmaları için değiştirilmesini garanti eder.
Tam olabilirlik yöntemleri, birkaç yayınlanmış çalışmada 10.000'den fazla lokus analiz edilmiş olsa da, veriler birkaç yüz lokustan oluştuğunda sınırlarına ulaşma eğilimindedir.
Uzantılar
Temel çoklu türlerin birleşmesi modeli, biyolojik üreme ve sürüklenme sürecinin ana faktörlerini barındırmak için çeşitli şekillerde genişletilebilir. Örneğin, sürekli zamanlı geçişin dahil edilmesi, geçişli yalıtım veya IM modelleri olarak da bilinen MSC+M (geçişli MSC için) modeline yol açar. Epizodik hibridizasyon/introgresyonun dahil edilmesi, introgresyon (MSci) veya çoklu tür-ağ-birleştirici (MSNC) modeliyle MSC'ye yol açar.
filogenetik tahmin üzerindeki etkisi
Çoklu türlerin birleşmesi, moleküler filogenetik teori ve pratiği için derin etkilere sahiptir. Tek tek gen ağaçları tür ağacından farklı olabileceğinden, tek bir lokus için ağaç tahmin edilemez ve gen ağacının tür ağacına karşılık geldiği varsayılamaz. Aslında, herhangi bir makul sayıda takson düşünüldüğünde, herhangi bir bireysel gen ağacının tür ağacından en azından bazı ilişkiler için farklı olacağından neredeyse emin olunabilir. Bununla birlikte, gen ağacı-tür ağacı uyumsuzluğunun, tür ağacını tahmin etmek için tek bir gen ağacının kullanılamayacağı basit gözleminin ötesine geçen tür ağacı tahmininin teori ve pratiği üzerinde bir etkisi vardır, çünkü parametre uzayının bir kısmı vardır. sık gen ağacı tür ağacıyla uyumsuzdur. Parametre uzayının bu kısmına anomali bölgesi ve gen ağacından daha sık ortaya çıkması beklenen uyumsuz gen ağaçları denir . tür ağacına uyanlara anormal gen ağaçları denir .
Anormallik bölgesinin varlığı, çok sayıda gen ağacının basitçe tahmin edilemeyeceğini ve en fazla sayıda gen ağacının tür ağacı olduğunu varsayamayacağını ima eder. Tabii ki, tür ağacını gen ağaçlarının "demokratik oyu" ile tahmin etmek, mümkün olan son derece fazla sayıda filogenetik ağaç göz önüne alındığında, anomali bölgesi dışındaki sınırlı sayıda takson için işe yarayacaktır. Bununla birlikte, anormal gen ağaçlarının mevcudiyeti, çoğunluk kuralı genişletilmiş ("açgözlü") konsensüs yöntemi veya cimri (MRP) süper ağaç yaklaşımı ile matris temsili gibi gen ağaçlarını birleştirmek için basit yöntemlerin tutarlı tahmin edici olmayacağı anlamına gelir . tür ağacı (yani yanıltıcı olacaktır). Yeterli sayıda gen ağacı kullanıldığı sürece, gen ağaçlarının en az %50'sinde bulunan grupların korunduğu gen ağaçları için basitçe çoğunluk kuralı konsensüs ağacının oluşturulması yanıltıcı olmayacaktır. Bununla birlikte, bir dizi gen ağacı için çoğunluk kuralı konsensüs ağacının yanlış kladlardan kaçınmak için bu yeteneği, çözülmemiş gruplara sahip olma pahasına gelir.
Simülasyonlar, tür ağacı parametre uzayının , filogeninin maksimum olabilirlik tahminlerinin, analiz edilen veri miktarı arttıkça artan olasılıkla yanlış ağaçlar olduğu bölümleri olduğunu göstermiştir. Bu önemlidir, çünkü farklı lokuslardan çoklu dizi hizalamalarının daha sonra maksimum olabilirlik (veya Bayesian MCMC ) analizi için kullanılan tek bir büyük süpermatris hizalaması oluşturmak üzere bir araya getirildiği "birleştirme yaklaşımı" hem uygulanması kolaydır hem de ampirik olarak yaygın olarak kullanılır. çalışmalar. Bu, bir model yanlış tanımlama durumunu temsil eder, çünkü birleştirme yaklaşımı örtük olarak tüm gen ağaçlarının aynı topolojiye sahip olduğunu varsayar. Gerçekten de, birleştirilmiş bir verinin maksimum olabilirlik analizi kullanılarak çoklu türlerin birleştirilmesi altında oluşturulan veri analizlerinin, analiz için kullanılan lokus sayısı arttıkça gerçek tür ağacında yakınsamasının garanti edilmediği kanıtlanmıştır (yani, maksimum olabilirlik birleştirme). istatistiksel olarak tutarsızdır).
Çoklu türlerin birleşmesi altında çıkarım için yazılım
Çok türlü kaynaşma çerçevesinde filogenetik tahmin için iki temel yaklaşım vardır: 1) hem maksimum olabilirlik hem de Bayes yöntemleri dahil olmak üzere, doğrudan çok noktalı dizi hizalamaları üzerinde çalışan tam olabilirlik veya tam veri yöntemleri ve 2) bir özet kullanan özet yöntemleri özet girdi olarak tahmini gen ağaçlarını kullanan iki aşamalı yöntemler ve özet girdi olarak lokuslar üzerinde havuzlanmış site deseni sayımlarını kullanan SVDQuartets dahil olmak üzere orijinal dizi verilerinin.
| programı | Açıklama | Yöntem | Referanslar |
|---|---|---|---|
| ASTRAL | ASTRAL (Doğru Türler TRee Algoritması), bir dörtlü yöntemi kullanarak bir dizi gen ağacını özetler ve birleşik dal uzunlukları ve destek değerleriyle tür ağacının bir tahminini oluşturur (yerel sonsal olasılıklar) | Özet | Mirarab et al. (2014); Zhang et al. (2018) |
| ASTRİD | ASTRID (Düğümler Arası Mesafelerden Doğru Tür Ağaçları), NJst yönteminin bir uzantısıdır. ASTRID/NJst, bir dizi girdi gen ağacından düğümler arası mesafeleri hesaplayan bir özet tür ağacı yöntemidir. Daha sonra tür ağacını bu mesafelerden tahmin etmek için komşu birleştirme veya minimum evrim gibi bir mesafe yöntemi kullanılır. ASTRID/NJst'nin bir eksik veri modeli altında tutarlı olmadığını unutmayın. | Özet | Vachaspati ve Warnow (2015) |
| BPP | Çok türlü birleşme süreci altında popülasyonlar arasında soyoluş ve ayrışma sürelerini çıkarmak için Bayesian MCMC yazılım paketi; ayrıca türlerin sınırlandırılması için bir yöntem içerir | Tam olasılık | Yang et al. (2015); Flouri et al. (2018) |
| STACEY | Çok türlü birleşme süreci altında popülasyonlar arasında soyoluş ve ayrışma sürelerini çıkarmak için Bayesian MCMC yazılım paketi; minimal kümeler (modele göre aynı türe ait olduğu varsayılan örnekler) MCMC sırasında parametre alanını değiştirmeye gerek kalmadan örneklenir | Tam olasılık | Jones ve ark. (2015); Jones GR (2018) |
| *CANAVAR | Çok türlü birleşme süreci altında popülasyonlar arasında soyoluş ve ayrışma sürelerini çıkarmak için Bayesian MCMC yazılım paketi. BEAST yazılım paketinin bir parçası olarak uygulandı (Star BEAST olarak telaffuz edilir) | Tam olasılık | Heled ve Drummond (2010) |
| MP-EST | Girdi olarak bir dizi gen ağacını kabul eder ve tür ağacının maksimum sözde-olabilirlik tahminini üretir. | Özet | Liu et al. (2010) |
| SVDquartets ( PAUP* ile uygulanır ) | PAUP*, birçok yöntemi uygulayan genel bir filogenetik tahmin paketidir. SVDquartets, çoklu türlerin birleşmesi göz önüne alındığında oluşturulan veriler için istatistiksel olarak tutarlı olduğu gösterilen bir yöntemdir. | Özet/Site modeli yöntemi | Chifman ve Kubatko (2014) |






























![{\displaystyle \exp\{-{\frac {n(n-1)}{\teta }}[\tau -(t_{m}+t_{m-1}+\ldots +t_{n+1} )]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/2f435c3e48b6ad522af7d79165c8713ab6a42446)







![{\displaystyle \prod _{j=n+1}^{m}{\Big [}{\frac {2}{\theta }}\exp {\Big \{}-{\frac {j(j-) 1)}{\theta }}t_{j}{\Big \}}{\Big ]}\exp {\Big \{}-{\frac {n(n-1)}{\theta }}(\ tau -(t_{m}+t_{m+1}+\ldots +t_{n+1})){\Big \}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/a7937f1541db74c1d61321eb52a49d5a32f19407)
![{\displaystyle {\begin{aligned}f(G_{i}\mid \Theta )&=[2/\theta _{H}\exp\{-6t_{3}^{(H)}/\theta _ {H}\}\exp\{-2(\tau _{HC}-t_{3}^{(H)})/\theta _{H}\}]\\&{}\kez [2/ \theta _{C}\exp\{-2t_{2}^{(C)}/\theta _{C}\}]\\&{}\times [2/\theta _{HC}\exp\ {-6t_{3}^{HC}/\theta _{HC}\}]\times [2/\theta _{HC}\exp\{-2t_{2}^{HC}/\theta _{HC }\}]\\&{}\times [\exp\{-2(\tau _{HCG}-\tau _{HG}-(t_{3}^{HC}+t_{2}^{HC) }))/\theta _{HCG}\}]\\&{}\kez [2/\theta _{HCGO}\exp\{-6t_{3}^{HCGO}/\theta _{HCGO}\ }]\times [2/\theta _{HCGO}\exp\{-2t_{2}^{HCGO}/\theta _{HCGO}\}]\end{aligned}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/2d396905de66e7b66c05298abe56ddc5be144a53)

















